Laboratorium Epitranskryptomiki

29 09 2022
O Grupie:
Wirusy stanowią jedno z największych zagrożeń dla zdrowia ludzkiego oraz globalnej gospodarki. Choć stosunkowo dobrze rozumiemy ich wpływ na zdrowie człowieka, nasza wiedza o interakcjach gospodarz–patogen u innych ssaków i zwierząt jest ograniczona. Różnice ewolucyjne oraz zróżnicowane nisze ekologiczne zasiedlane przez różne gatunki sprawiają, że niemal niemożliwe jest bezpośrednie przeniesienie wiedzy o danym procesie z komórek ludzkich na inne zwierzęta.
Biorąc pod uwagę, że zwierzęta hodowlane stanowią kluczowe źródło białka dla ludzi, zrozumienie, w jaki sposób zwierzęta bronią się przed patogenami, ma zasadnicze znaczenie dla utrzymania globalnego bezpieczeństwa żywnościowego. Co więcej, wirusy wywołujące choroby zakaźne u zwierząt hodowlanych mogą łatwo przenieść się na nowego gospodarza, np. ludzi, i przyczynić się do wywołania epidemii czy nawet pandemii.
Badania prowadzone w Laboratorium Epitranskryptomiki mają na celu zrozumienie, w jaki sposób wrodzona odporność immunologiczna pomaga w zwalczaniu infekcji wywołanych wirusami RNA, do których zaliczają się wirus grypy, SARS-CoV-2, wirus Dengi czy wirus Eboli. Nasze badania koncentrują się na poznaniu, w jaki sposób receptory komórek gospodarza rozpoznają wirusowe RNA, a także jak wirusy maskują swoje RNA, aby uniknąć rozpoznania przez system obronny komórek.
Działalność badawcza:
Realizowane projekty:
- Zrozumienie jak wirusowy epitranskryptom kształtuje odpowiedź immunologiczną gospodarza; finansowanie: Narodowe Centrum Nauki, Sonata Bis (2022-2026), kierownik: Paweł Sikorski;
- Nowe wszechstronne narzędzie do znakowania RNA; fnansowanie: IDUB Nowe Idee (2023-2024), kierownik: Paweł Sikorski;
- FLU-SWITCH Identification of factors driving the emergence and spread of avian influenza viruses with zoonotic potential; projekt koordynowany przez dr. Romaina Volmera, INRAE; polski partner: Paweł Sikorski;
ERA-NET ICRAD (International Coordination of Research on Infectious Animal Diseases) (2023-2026).
Publikacje:
-
- Drazkowska K., Cieslicka J., Kitowicz M., Pastucha A., Markiewicz L., Szymanek W., Goryca K., Kowalczyk T., Cysewski D., Bausch A.R., Sikorski P.J. (2025) Effective recognition of double-stranded RNA does not require activation of cellular inflammation. Science Advances, 11(15).
- Gackowska K., Krejmer-Rabalska M., Drazkowska K., Jemielity J., Krol E., Szewczyk B. (2025) Affinity-purified sHBsAg-based virus-like particles as a platform for foreign mRNA binding. Virology, 110651.
- Drazkowska K., Tomecki R., Tudek A. (2025) Purification of Enzymatically Active Xrn1 for Removal of Non-capped mRNAs from In Vitro Transcription Reactions and Evaluation of mRNA Decapping Status In Vivo. Methods in Molecular Biology. 2863: 81-105.
- Tomecki R., Drazkowska K., Madaj R., Mamot A., Dunin-Horkawicz S., Sikorski P.J. (2024) Expanding the Available RNA Labeling Toolbox With CutA Nucleotidyltransferase for Efficient Transcript Labeling with Purine and Pyrimidine Nucleotide Analogs. ChemBioChem. 25(15): e202400202
- Warminski M., Trepkowska E., Smietanski M., Sikorski P.J., Baranowski M.R., Bednarczyk M., Kedzierska H., Majewski B., Mamot A., Papiernik D., Popielec A., Serwa R.A., Shimanski B.A., Sklepkiewicz P., Sklucka M., Sokolowska O., Spiewla T., Toczydlowska-Socha D., Warminska Z., Wolosewicz K., Zuberek J., Mugridge J.S., Nowis D., Golab J., Jemielity J., Kowalska J. (2024) Trinucleotide mRNA Cap Analogue N6-Benzylated at the Site of Posttranscriptional m6Am Mark Facilitates mRNA Purification and Confers Superior Translational Properties In Vitro and In Vivo. Journal of the American Chemical Society. 146(12): 8149-8163.
- Tomecki R., Drazkowska K., Kobylecki K., Tudek A. 2023. SKI complex: A multifaceted cytoplasmic RNA exosome cofactor in mRNA metabolism with links to disease, developmental processes, and antiviral responses. Wiley Interdisciplinary Reviews RNA. e1795.
- Drazkowska K., Tomecki R., Warminski M., Baran N., Cysewski D., Depaix A., Kasprzyk R., Kowalska J., Jemielity J., Sikorski P.J. 2022. 2′-O-Methylation of the second transcribed nucleotide within the mRNA 5′ cap impacts the protein production level in a cell-specific manner and contributes to RNA immune evasion. Nucleic Acids Research. 50: 9051–9071.
Aparatura badawcza:
Kierownik zespołu:
Dr hab. Paweł Sikorski pracę doktorską obronił na Wydziale Biologii UW (promotor – prof. Joanna Kufel). Następnie odbył staż podoktorski w grupie prof. Jacka Jemielitego w Centrum Nowych Technologii UW. Od 2022 roku pracuje jako adiunkt na Wydziale Biologii UW oraz kieruje Laboratorium Epitranskryptomiki. W 2023 otrzymał stopnień doktora habilitowanego, za osiągnięcia naukowe składające się na rozprawę habilitacyjną otrzymał Nagrodę Jego Magnificencji Rektora UW oraz Nagrodę Prezesa Rady Ministrów.
W trakcie swojej pracy naukowej odbył staż zagraniczny w laboratorium dr Dominique Gagliargiego (IBMP, Strasburg, Francja).
Ponadto, jest autorem ponad 20 publikacji naukowych w czasopismach takich jak Nucleic Acids Research, Journal of the American Chemical Society czy Angewandte Chemie oraz 4 międzynarodowych zgłoszeń patentowych.
Szczególne osiągnięcie:
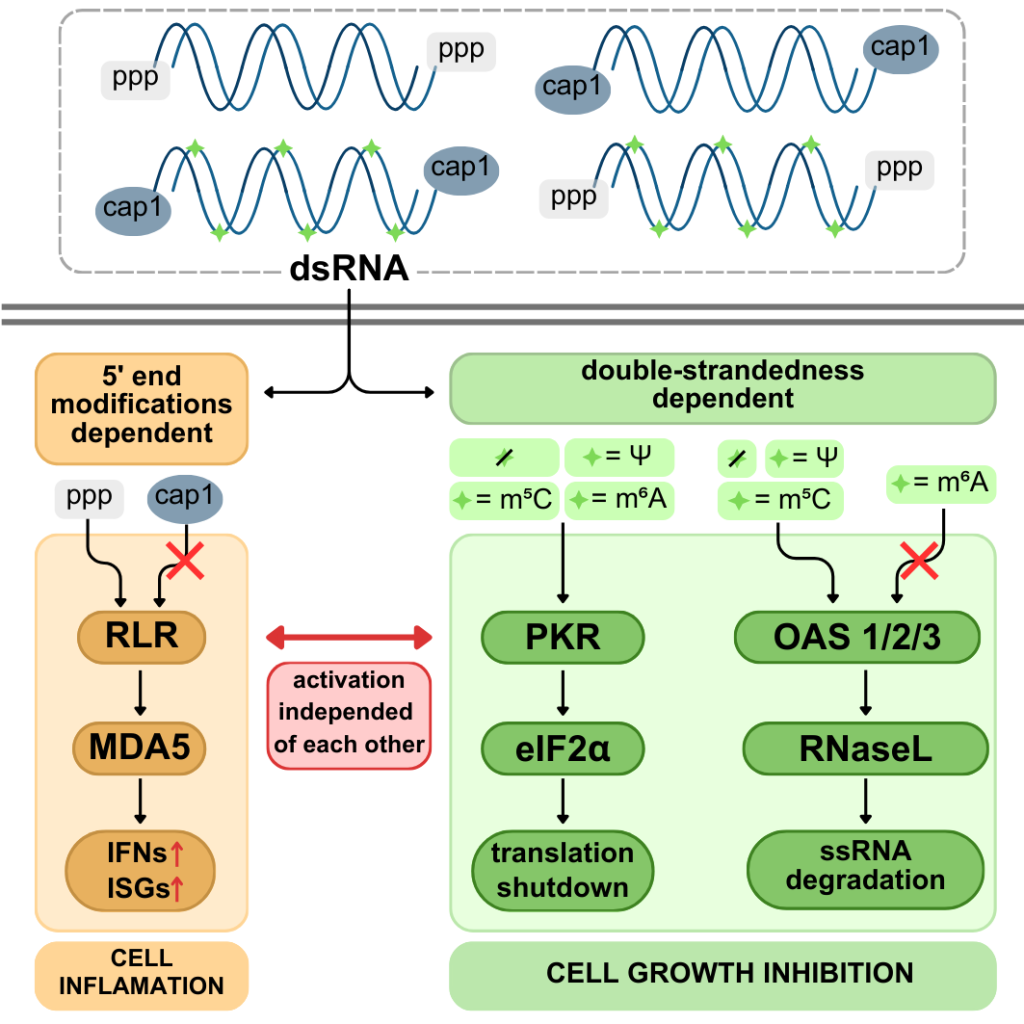
Nadmiar dwuniciowego RNA (dsRNA) w ludzkich komórkach, często pojawiający się w wyniku infekcji wirusowych, aktywuje wrodzone szlaki odpornościowe wywołujące stan zapalny i hamujące wzrost komórek. Ostatnio pokazaliśmy, że odpowiedź na dsRNA może zachodzić bez wywołania stanu zapalenego. Prozapalny szlak RLR oraz szlaki hamujące wzrost (OAS/RNaza L i PKR) działają niezależnie – końce 5’ dsRNA indukują stan zapalny, podczas gdy struktury dupleksowe RNA aktywują OAS/RNazę L i PKR.
Co zaskakujące, obecność modyfikacji RNA, takich jak N6-metyloadenozyna (m6A), 5-metylocytozyna i pseudourydyna, nie ma większego wpływu na immunogenność dsRNA. Okazało się jednak, że m6A specyficznie hamuje aktywację szlak OAS/RNase L. Nasze badania pokazują, jak precyzyjnie wrodzona odporność dostosowuje się do konkretnych zagrożeń.