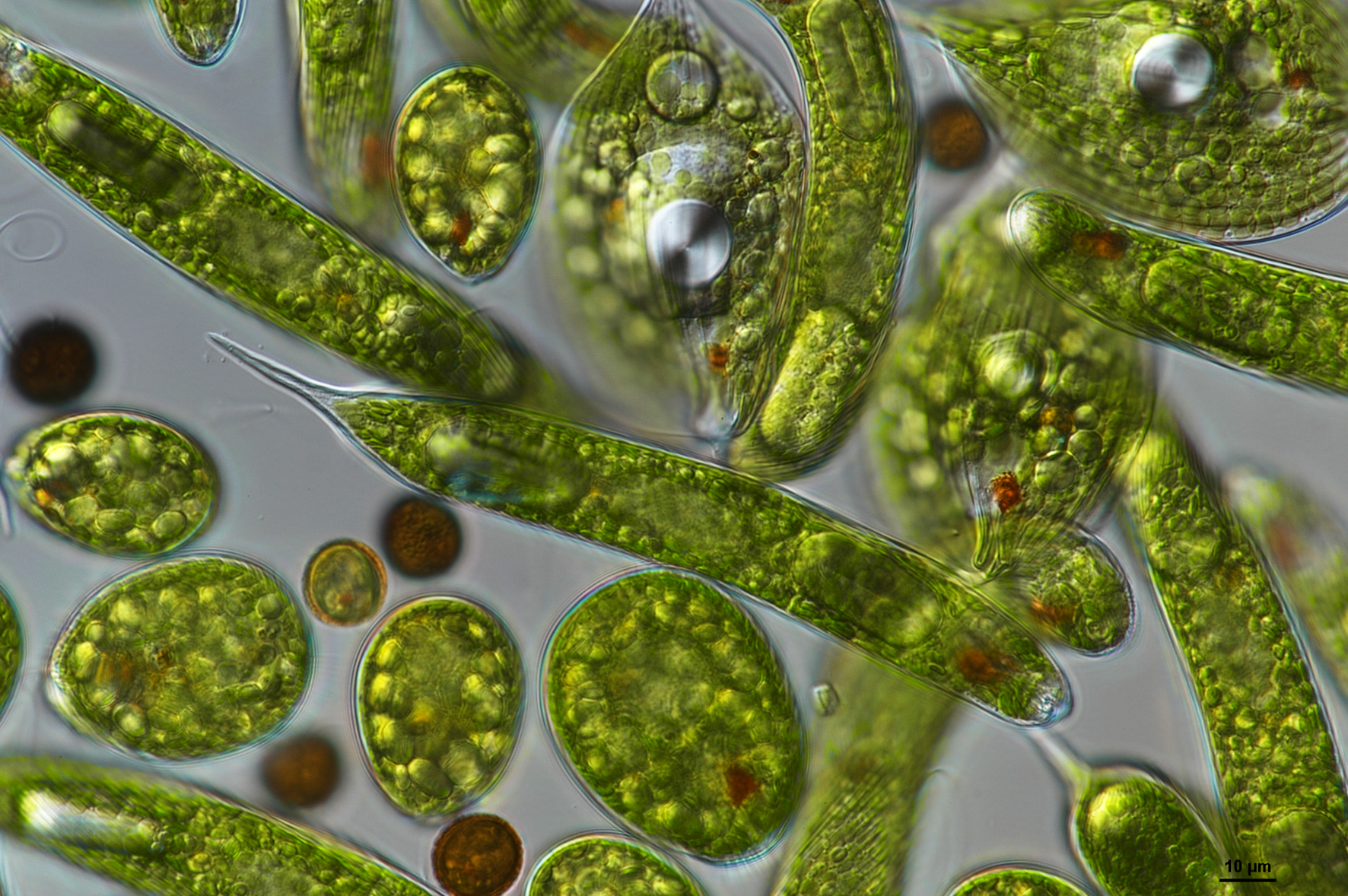
Biologia mikroorganizmów eukariotycznych
O Grupie:
Badania wykonywane przez grupę dotyczą różnorodności, ekologii i ewolucji protistów, czyli jednokomórkowych eukariontów. Badania opierają się o pracę terenową, hodowle, analizy molekularne, mikroskopię oraz bioinformatykę. Grupa badawcza używa metod mikroskopowych i molekularnych, aby przyjrzeć się różnym grupom protistów, zbadać ich różnorodność i występowanie oraz rozwikłać ich relacje pokrewieństwa i taksonomię. Sekwencjonowanie genomów i transkryptomów umożliwia badania nad ewolucję genomu jądrowego oraz genomów organellarnych, ewolucją splicingu i interakcji endosymbiotycznych. Badają zarówno jednokomórkowe glony – w szczególności eugleniny, protisty heterotroficzne, jak i pasożyty. W ramach grupy pracują trzy zespoły koncentrujące się na różnych aspektach biologii mikroorganizmów eukariotycznych.
Więcej informacji o grupie badawczej: https://ibe.biol.uw.edu.pl/grupy-badawcze/biologia-mikroorganizmow-eukariotycznych/
Działalność badawcza:
• Różnorodność i interakcje mikroorganizmów eukariotycznych i bakterii w wodach słodkich.
Celem projektu jest identyfikacja mikroorganizmów eukariotycznych i bakterii występujących w jeziorach Pojezierza Mazurskiego z wykorzystaniem metod molekularnych. Analiza współwystępowania protistów i bakterii umożliwi identyfikację ich interakcji. Szczególnie interesują nas interakcje protistów i patogennych bakterii, co pomoże określić naturalny rezerwuar tych bakterii. Badania w jeziorach o różnym statusie troficznym umożliwią natomiast ocenę wpływu użyźnienia jezior na skład zespołów mikroorganizmów, przewidzieć zmiany w ekosystemach słodkowodnych wynikające z działalności człowieka i przyczyni się do ich skuteczniejszej ochrony.
• Różnorodność i ewolucja interakcji endosymbiotycznych u mikroorganizmów eukariotycznych.
Pracujemy nad zrozumieniem jak powstaje relacja endosymbiotyczna, poprzez identyfikację i charakteryzację nowych układów protisty-endosymbionty. Poszukujemy takich interakcji w środowiskach wodnych izolując i identyfikując partnerów metodami mikroskopowymi i genetycznymi. Badamy ultrastrukturę, ewolucję genomu i regulację ekspresji genów, które towarzyszą interakcji endosymbiotycznej. Wyniki badań pozwolą na głębsze zrozumienie jednego z procesów ewolucyjnych, który doprowadził do powstania złożonych komórek eukariotycznych. Poznanie różnorodności endosymbioz przyczyni się także do zrozumienia roli interakcji mikroorganizmów eukariotycznych w funkcjonowaniu ekosystemów wodnych.
• Ewolucja genomów jądrowych euglenin.
Genomy jądrowe w tej grupy organizmów są niespodziewanie duże, o wysokim stopniu powtarzalności i niestandardowej strukturze. Co więcej, w genach jądrowych, oprócz konwencjonalnych intronów spliceosomalnych obecnych u większości eukariontów, występują także introny niekonwencjonalne, usuwane najprawdopodobniej niezależnie od standardowego spliceosomu. Badamy pochodzenie intronów niekonwencjonalnych, mechanizm ich usuwania z pierwotnych transkryptów i mechanizmy ich wstawiania w nowych pozycjach w genach jądrowych. Zajmujemy się także ewolucją i wpływem na biologię euglenin splice-liderowego trans-splicingu (SLTS), procesu, w którym do cząsteczek mRNA dołączane są identyczne sekwencje liderowe.
• Taksonomia, filogeneza, ewolucja i identyfikacja zielonych euglenin.
Staramy się lepiej poznać różnorodność euglenin poprzez izolowanie nowych gatunków ze środowiska, obserwacje mikroskopowe i analizy filogenetyczne pozwalają odtworzyć relacje pokrewieństwa między nimi. Opracowaliśmy również uniwersalną, szybką i dokładną metodę identyfikacji molekularnej taksonów na podstawie barkodów DNA, która umożliwi monitoring zbiorników wodnych pod kątem bioróżnorodności euglenin, w tym gatunków tworzących toksyczne zakwity.
• Aplikacyjne zastosowania zielonych eulgenin.
Eugleniny nie tylko dobrze tolerują obecność wysokich stężeń metali ciężkich i różnych substancji toksycznych w środowisku, ale są również zdolne do ich kumulacji, co można wykorzystać w biologicznym oczyszczaniu wód zanieczyszczonych ściekami przemysłowymi. Zawierają również wysoce aktywny biologicznie matriał zapasowy 1-3 β glukan. Pracujemy nad selekcją gatunków euglenin, z któwych wydajnie można pozyskiwać 1-3 β glukan.
| Nazwa aparatu | Kontakt do operatora/pokój | |
| 1 | MinION – zestaw do sekwencjonowania nanoporowego | dr Karolina Grabowska-Grucza, pok.: 4.145, e-mail: k.grabowska7@uw.edu.pl |
| 2 | System Agilent TapeStation do automatycznej elektroforezy i kontroli jakości próbek DNA/RNA | dr Karolina Grabowska-Grucza, pok.: 4.145, e-mail: k.grabowska7@uw.edu.pl |
| 3 | Mikroskop fluorescencyjny Nikon Ti2-E z kamerą | mgr Alicja Fells, pok.: : 4.148, e-mail: a.fells@uw.edu.pl |
Kierownik zespołu:
Dr hab. Anna Karnkowska, prof. ucz. jest pracownikiem Wydziału Biologii Uniwersytetu Warszawskiego. Specjalizuje się w badaniach różnorodności, ewolucji i ekologii mikroorganizmów eukariotycznych, od glonów po pasożyty. Odbyła dwa długoterminowe staże podoktorskie na Uniwersytecie Karola w Pradze (Czechy) oraz na Uniwersytecie Kolumbii Brytyjskiej w Vancouver (Kanada) i liczne krótsze wizyty w tym na Uniwersytecie Alberty (Kanada), w Instytucie Biologii Ewolucyjnej w Barcelonie (Hiszpania) i w stacji badawczej Uniwersytetu Sorbońskiego w Roscoff (Francja). Jest członkinią zarządu i w roku 2023/2024 pełni rolę prezydenta międzynarodowego towarzystwa protistologicznego (ISOP). W 2023 roku została wybrana na członkinię Europejskiej Akademii Mikrobiologii.
W swoim dorobku ma ponad 50 publikacji naukowych, a także cztery rozdziały w książkach. Na swoje badania uzyskała EMBO Installation Grant a także cztery granty NCN. Za swoją działalność naukową uzyskała liczne wyróżnienia, w tym stypendium START Fundacji na Rzecz Nauki Polskiej (2013), Stypendium Ministerstwa Nauki i Szkolnictwa Wyższego dla wybitnych młodych naukowców (2018), Nagrodę im. Prof. Pieńkowskiego (2020) i Stypendium 20. edycji programu L’Oréal-UNESCO dla Kobiet i Nauki (2021).
Szczególne osiągnięcie:
Zespół wniósł znaczący wkład w mikrobiologię Eukariontów wykorzystując protisty jako modele do badania powstawania, ewolucji i utraty organelli takich jak chloroplasty i mitochondria. Badania pogłębiły również naszą wiedzę na temat różnorodności, filogenezy i biologii molekularnej tych nadal słabo poznanych organizmów o rosnącym potencjale biotechnologicznym. Ponadto zespół opracował nowe narzędzia bioinformatyczne do analizy danych z sekwencjonowania wysokoprzepustowego, które przyczynią się do dalszego postępu w badaniach różnorodności, ekologii i ewolucji mikroorganizmów eukariotycznych.